D'après les travaux de François Ferron, chercheur CNRS au laboratoire Architecture et fonction des macromolécules biologiques et Isabelle Imbert, enseignante-chercheuse d'Aix-Marseille Université au laboratoire Architecture et fonction des macromolécules biologiques (CNRS/Aix-Marseille Université)
Quand un virus à ARN devient fidèle : vers de nouvelles stratégies de lutte contre les coronavirus
Par Rédaction - Théragora
Théragora - www.theragora.fr - Année 2017 - CNRS N° 12 - Page 0 - crédits iconographique François Ferron
Des chercheurs du CNRS et d’Aix Marseille Université ont reconstitué in vitro la machinerie permettant au SARS-Coronavirus de corriger les erreurs faites au cours de la réplication de son énorme génome d’ARN (> 30-kb). De façon similaire aux organismes à ADN, ces virus ont acquis une activité exonucléase qui s’associe à l’ARN polymérase virale, pour exciser les mésappariements. Ce système, porteur également d’une nouveauté structurale, va aider à l’identification d’inhibiteurs contre cette famille de virus. En effet, il n’existe aucun traitement contre les infections à Coronavirus alors qu’ils sont une réelle menace vis à vis de la santé humaine. Ces travaux sont publiés dans la revue PNAS.
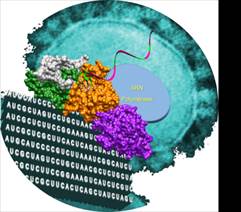
La structure du complexe de réplication du virus du SARS - La forme bleue représente l’enzyme qui recopie le génome du virus (ARN polymérase). Le système de contrôle qualité est représenté en vert, orange et blanc. Un ARN partiellement double brin a été positionné dans la poche catalytique de l’exonucléase, l’enzyme qui corrige les erreurs (en vert et orange).
Les coronavirus (CoV) sont des virus ubiquitaires infectant une très grande variété d’hôtes. Chez l’homme, ils étaient essentiellement responsables d’atteintes bénignes du tractus respiratoire. En 2003, un nouveau coronavirus a émergé et a été responsable de l’épidémie mondiale du Syndrome Respiratoire Aigu Sévère (SARS), une forme très dangereuse de pneumonie. Pendant la seule épidémie de SARS, plus de 8000 personnes ont été infectées parmi lesquelles 900 ont péri (soit un taux de mortalité de 10%), dans ce qui a été la première pandémie du XXIème siècle. Quasiment 10 ans plus tard, fin 2012, l’histoire se répète avec l’émergence d’un nouveau CoV, le MERS-CoV (pour Middle East Respiratory Syndrome-CoV) qui a infecté jusqu’à maintenant plus de 2100 personnes et dont l’infection est associée à un taux de mortalité d’environ 35%. A l’heure actuelle, il n’existe aucun traitement contre les coronavirus. De façon surprenante, la ribavirine qui est un antiviral à large spectre d’action n’a pas d’effet sur les patients infectés par le SARS-CoV ou le MERS-CoV.
Le génome des coronavirus est un ARN simple brin de polarité positive, c’est à dire qu’il est directement traduit par les ribosomes cellulaires, et est le plus long des virus à ARN ( 30 000 nucléotides).
Les chercheurs du laboratoire AFMB à Marseille ont d’une part reconstitué in vitro le complexe multi-protéique qui permet de produire de multiples copies du génome du SARS-CoV. Ils ont pu montrer que l’ARN polymérase ARN-dépendante virale, seule, commettait beaucoup d’erreurs (non-respect des appariements Watson-Crick) lorsqu’elle copiait un ARN matrice. Cette faible fidélité, incompatible avec la survie du virus, a été compensée par l’acquisition au cours de l’évolution, d’une activité exonucléase. Ainsi, en rajoutant cette activité de correction des erreurs ou « proofreading », les chercheurs ont pu montrer la correction d’un mésappariement. Avec ce « mini-complexe » de réplication, ils ont pu également montrer que même si la polymérase virale est capable d’incorporer la ribavirine, l’inhibiteur est excisé par l’activité exonucléase. Ainsi, pour la première fois, une explication est avancée quant à l’inefficacité de ce médicament contre les coronavirus.
D’autre part, la protéine portant l’activité exonucléase possède un second domaine qui permet la méthylation de la coiffe des ARN messagers viraux. L’équipe a mis en évidence que ces 2 activités enzymatiques sont régulées par des mouvements de grandes amplitudes, entrainant des modifications structurales de la protéine. Contre toute attente, le domaine qui méthyle la coiffe des ARNm viraux possède un repliement unique. Ce nouveau repliement diffère énormément du seul repliement qui avait été décrit jusqu’alors. Cette découverte implique que les coronavirus ont créé ce domaine et non pas « volé » un gène déjà existant comme c’est souvent le cas au cours de l’évolution.
Cette étude structure/fonction montre que les coronavirus se situent à un des points de bascule de l’évolution pour la stabilité génomique. De plus, cette découverte contribue au développement de nouvelles stratégies anti-coronavirus.
Référence :
Structural and molecular basis of mismatch correction and ribavirin excision from Coronavirus RNA, François Ferron, Lorenzo Subissi, Ana Theresa Silveira De Morais, Nhung Thi Tuyet Le, Marion Sevajol, Laure Gluais, Etienne Decroly, Clemens Vonrhein, Gérard Bricogne, Bruno Canard & Isabelle Imbert. PNAS, 25 décembre 2017. www.pnas.org/cgi/doi/10.1073/pnas.1718806115










